
3つの要点
✔️ヒトの遺伝学において、スプライシング(遺伝情報から不要な部分を取り除き再度つなぎ合わせる編集作業)による遺伝的変異の予測は非常に重要だ。
✔️「MMSplice」とは、ゲノムや遺伝子に関する大規模データを学習するニューラルネットワークである。
✔️「MMSplice」を用いると、スプライシングがヒトの遺伝的変異に与える影響を高精度で予測することができる。
詳細
スプライシングによる遺伝的変異の影響を予測することは、ヒトの遺伝学において非常に重要だ。
CAGI5エクソンスキップ予測の挑戦の上位モデルを構築したフレームワーク、MMSplice(スプライシングのモジュラーモデリング)について説明しよう。
MMSpliceモジュールは、エクソン、イントロン、およびスプライス部位をスコア付けするニューラルネットワークで、個別の大規模ゲノミクスデータセットについて学習する。
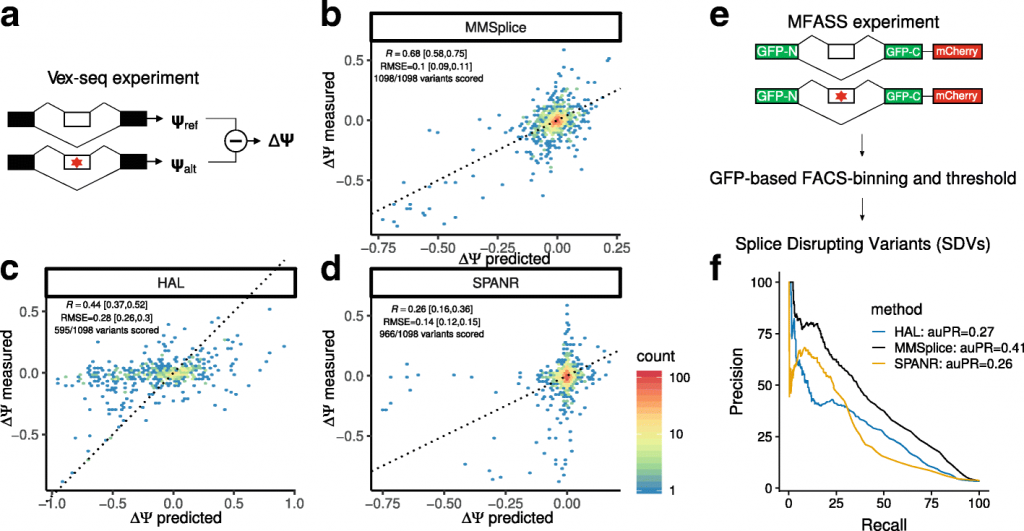
MMSpliceはエクソンスキッピングに対する変異効果の予測を改善する。
a Vex-seq実験のスキーマ。110の選択的エクソンからのまたは隣接する2059のExAC多様体(赤い星)の効果を、RNAseqによる参照配列(Ψref)および選択的(Ψalt)のパーセントスプライスインを測定することによってレポーター遺伝子で試験した。
b–d Vex-seq検定でのMMSplice(b)、HAL(c)、およびSPANR(d)の代替シーケンスと参照シーケンス間の測定値(y軸)と予測値(x軸)Ψの差データ。カラースケールは六角形の箱の中の数を表す。黒い線はy=xの対角線を表している。各プロットは、検討対象のモデルが採点できる多様体(バリアント;個体による遺伝的変異)の部分集合とともに表示される。ピアソン相関(R)および二乗平均平方根誤差(RMSE)もまた、スコア付けされた多様体に基づいて計算された。これら2つの測定基準の95%信頼区間は、ブートストラップで計算されている。
e MFASS実験の概要。設計された構築物のゲノム統合によって、2339のエクソンにまたがるかまたは隣接する27,733のExAC SNV(赤い星)のエクソンスキッピング効果を試験した。スプライス破壊多様体(SDV)は、元のエクソン包含指数が0.5以上であるエクソンを少なくとも0.5だけ変化させる多様体として定義される。
f モデル予測ΔΨに基づくMFASS SDV分類の精密想起曲線。3つのモデルすべての精度再現率曲線は、スコアを付けることができる多様体のセットに対して計算された。MMSplice(黒)は27,733の亜種すべてを獲得し、SPANR(黄色)は27,663の多様体(1,048 SDV)を獲得し、HAL(青)は14,353の多様体(489 SDV)を獲得した。
これらのモジュールを組み合わせることで、エクソンスキッピング、スプライス部位の選択、スプライシング効率、および病原性に対する変異の影響を、最先端技術と同等以上のパフォーマンスで予測する。
Kipoiのリポジトリで利用可能なモデルは、VCFファイルから直接インデルを含む亜種に適用される。
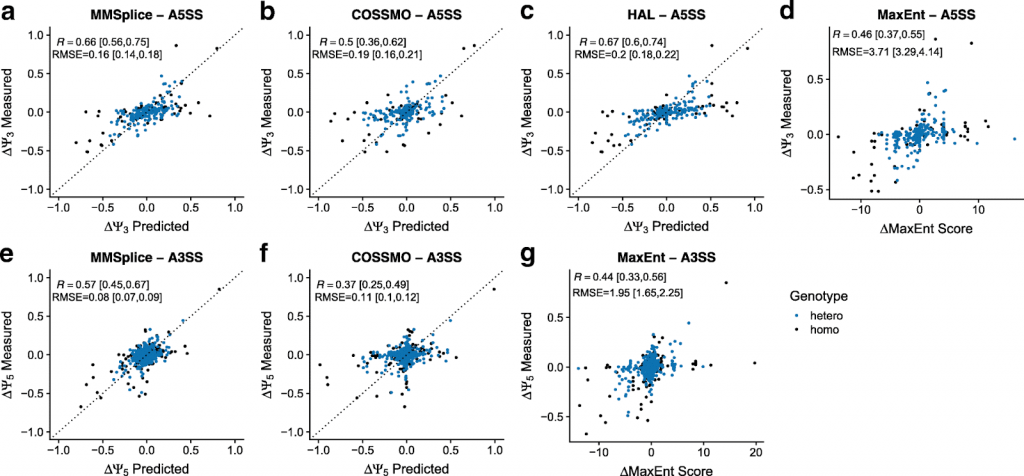
GTExデータセットでΔΨ5とΔΨ3を予測するモデルの評価。代替スプライスドナー(エクソン中3ntおよびイントロン中6nt)およびアクセプター(エクソン中3ntおよびイントロン中20nt)の周囲のGTEx多様体についての関連する効果(y軸)対予測(x軸)は以下の通りであった。同型(黒)および異型(青)の代替変種および同型の参照変種のΨ5(またはΨ3)は、脳および太陽に曝されていない皮膚サンプル上の同じ遺伝子型(スプライス部位300nt以内の複数の多様体をもつ個体を除く)を持つ個体の平均Ψ5(またはΨ3)を取ることによって計算された。ドナー多様体については、MMSplice(a)を、COSSMO(b)、HAL(c)、およびMaxEntScan(d)を基準にして評価した。アクセプター多様体については、MMSplice(e)を、COSSMO(f)およびMaxEntScan(g)を基準として評価した。ピアソン相関(R)および二乗平均平方根誤差(RMSE)についての95%信頼区間は、ブートストラップを用いて計算した。点線はy=xの対角線を表している。
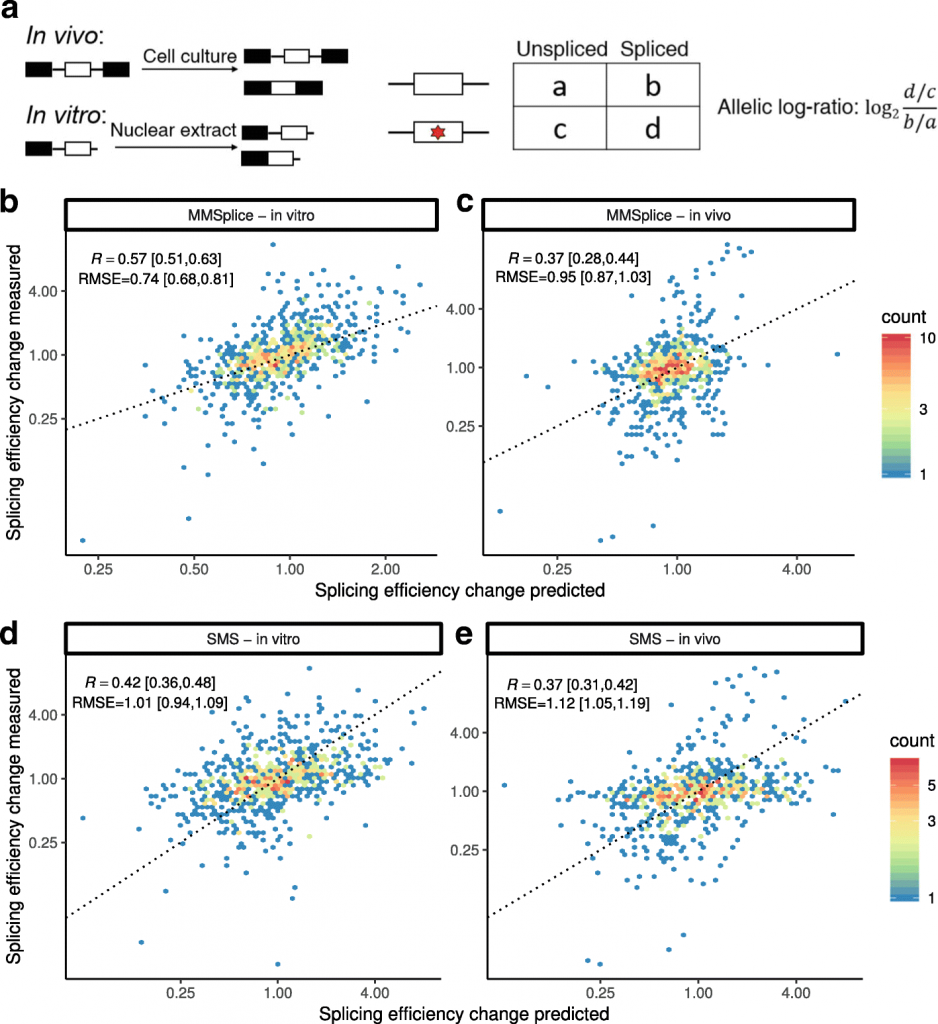
スプライシング効率の予測
a MaPSy実験。5761の公表された疾患を引き起こすエクソン変異のスプライシング効率に対する効果をインビボおよびインビトロの両方で測定する。スプライシング効率の変化を対立遺伝子対数比によって定量した。
b-e MMSplice(b、c)とSMSスコア(d、e)のテストセットにおける797の多様体についての対立遺伝子比の、測定値(y軸)対予測値(x軸)。点線はy=xの対角線を表す。ピアソン相関(R)および二乗平均平方根誤差(RMSE)についての95%信頼区間は、ブートストラップを用いて計算した。
この記事で取り扱った論文:J. Cheng, T. Y. D. Nguyen, K. J. Cygan, M. H Çelik, W. G. Fairbrother, Z. Avsec and Julien Gagneur, “MMSplice: modular modeling improves the predictions of genetic variant effects on splicing”. Genome Biology, 20, 48 (2019). [DOI: 10.1186/s13059-019-1653-z]
■サポートのお願い
AIDBを便利だと思っていただけた方に、任意の金額でサポートしていただけますと幸いです。







 PAGE TOP
PAGE TOP