(Featured AI and healthcare) Crohn Disease patients can be classified by machine learning! Which method is more effective? (Publication)
[論文] A. Romagnoni, S. Jégou, K. V. Steen, G. Wainrib, J.P. Hugot, International Inflammatory Bowel Disease Genetics Consortium, “Comparative performances of machine learning methods for classifying Crohn Disease patients using genome-wide genotyping data”. Scientific Reports 9, 10351 (2019).
[DOI: 10.1038/s41598-019-46649-z]
3つの要点
✔️機械学習(ML)を利用して、クローン病患者の分類を行った。
✔️分析には、ペナルティ付きロジスティック回帰(LR)、勾配ブースティング木(GBT)、人工ニューラルネットワーク(NN)といった手法が使われた。
✔️特に、GBTやNNのような非線形モデルは、遺伝子マーカーを同定・分類する有力なアプローチになると考えられる。
概説
クローン病(Crohn Disease;CD)は、ゲノムワイド関連解析(Genome Wide Association Studies;GWAS)を用いて140を超える遺伝子が同定されている複雑な遺伝的疾患である。
しかしながら、形質の遺伝的構造はほとんど知られていないままとなっている。
最近の機械学習(Machine Learning;ML)アプローチの発展により、研究者は、MLを用いて、健康な人と病気にかかった人を彼らのゲノム情報に従って分類するという着想を得た。
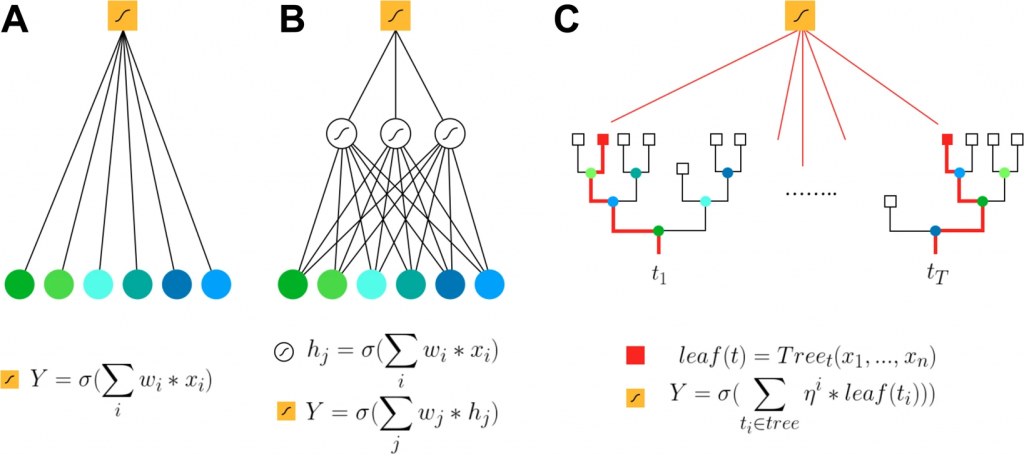
国際炎症性腸疾患ジェネティクス・コンソーシアム(International Inflammatory Bowel Disease Genetic Consortium;IIBDGC)に登録および遺伝子型決定された18,227人のCD患者および34,050人の健康な統制群を含むImmunochip(自己免疫疾患や炎症性疾患を詳細に解析するための分析器具)データセットは、ペナルティ付きロジスティック回帰(Logistic Regression;LR)、勾配ブースティング木(Gradient Boosted Trees;GBT)、人工ニューラルネットワーク(Neural Networks;NN)といった一連のML法を用いて再分析された。
各手法を比較するために使用された主なスコアは、ROC曲線下面積(Area Under the ROC Curve;AUC)統計である。
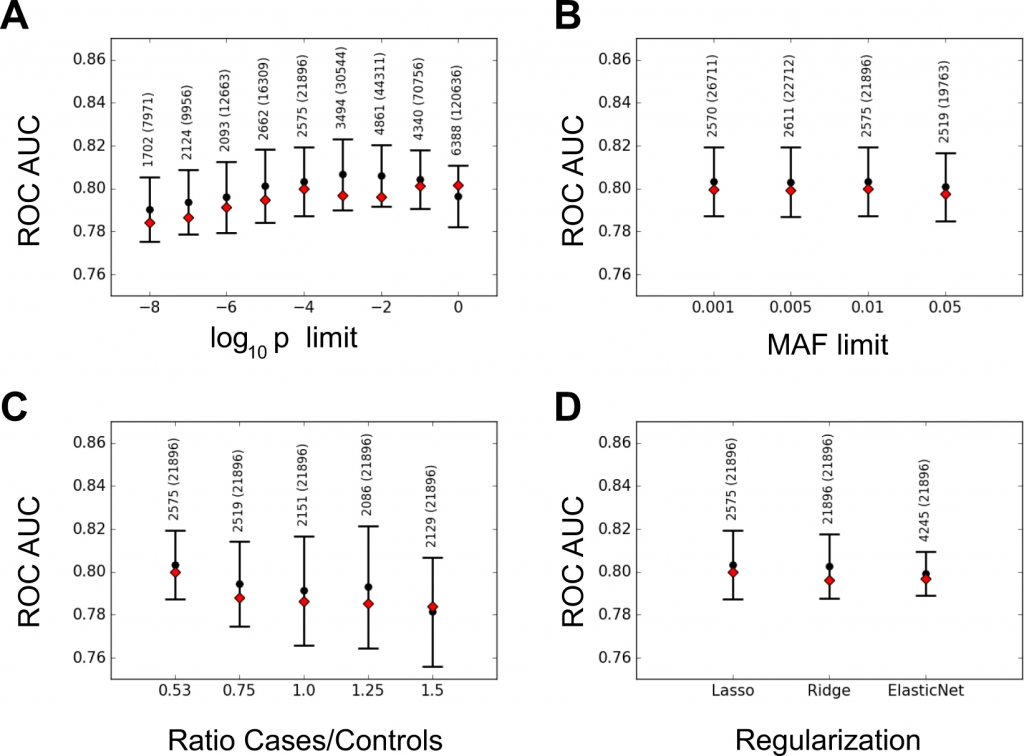
LRの結果に対する品質管理(Quality Control;QC)や入力およびコーディング手法の影響は、QC方法および欠けている遺伝子型の入力が人工的にスコアを増加させる可能性があることを示した。
反対に、患者/統制群比もマーカーの事前選択もコーディング戦略も結果に有意な影響を及ぼさなかった。
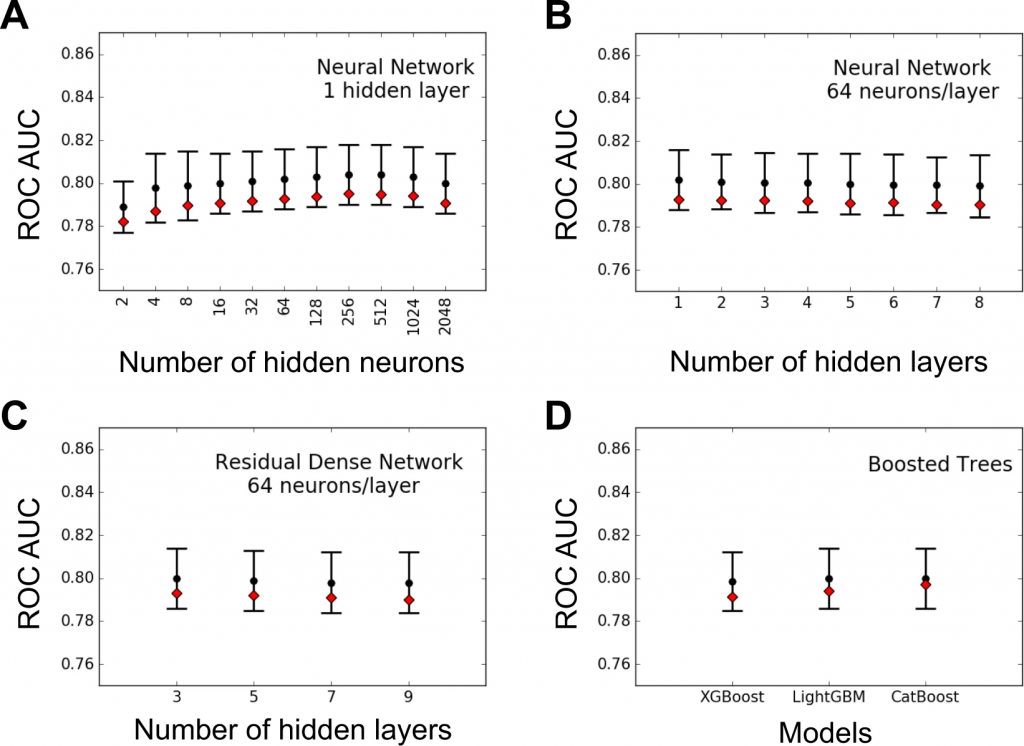
Lasso、Ridge、ElasticNetなどのLR法でも同様の結果が得られ、最大AUCは0.80であった。
XGBoost、LightGBM、CatBoostなどのGBTメソッドは、1つ以上の隠れ層を持つ密集したNNと共に類似のAUC値を示し、これは特性の遺伝的アーキテクチャにおける限られた上位効果を示唆している。
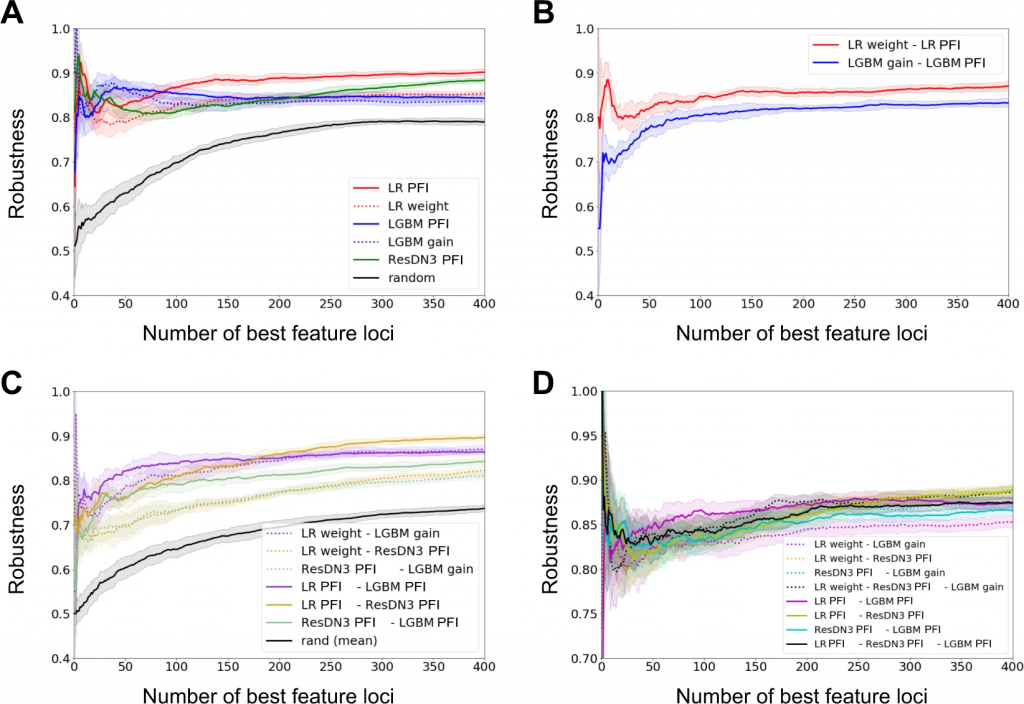
ML法は、最良の予測因子と効果の低い追加の予測因子の中で、これまでにGWASによって同定されたほぼすべての遺伝的変異型を検出した。
異なる手法の頑健性と相補性も研究されている。
LRと比較して、GBTまたはNNのような非線形モデルは、遺伝子マーカーを同定および分類するための強力な相補的アプローチを提供し得る。
著者
Alberto Romagnoni (Centre de recherche sur l’inflammation UMR 1149, Inserm – Université Paris Diderot, 75018, Paris, France / Data Team, Département d’informatique de l’ENS, École normale supérieure, CNRS, PSL Research University, 75005, Paris, France)
Simon Jégou (Owkin, 75011, Paris, France)
Kristel Van Steen (WELBIO, GIGA-R Medical Genomics – BIO3, University of Liège, Liège, Belgium / Department of Human Genetics, University of Leuven, Leuven, Belgium)
Gilles Wainrib (Data Team, Département d’informatique de l’ENS, École normale supérieure, CNRS, PSL Research University, 75005, Paris, France / Owkin, 75011, Paris, France)
Jean-Pierre Hugot (Centre de recherche sur l’inflammation UMR 1149, Inserm – Université Paris Diderot, 75018, Paris, France / Hôpital Robert Debré, Assistance Publique-Hôpitaux de Paris, 75019, Paris, France)
International Inflammatory Bowel Disease Genetics Consortium (IIBDGC)
出版情報
Received: 11 March 2019 / Accepted: 3 July 2019 / Published: 17 July 2019
Open Access This article is licensed under a Creative Commons Attribution 4.0 International License, which permits use, sharing, adaptation, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons license, and indicate if changes were made. The images or other third party material in this article are included in the article’s Creative Commons license, unless indicated otherwise in a credit line to the material. If material is not included in the article’s Creative Commons license and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder. To view a copy of this license, visit http://creativecommons.org/licenses/by/4.0/.
■サポートのお願い
AIDBを便利だと思っていただけた方に、任意の金額でサポートしていただけますと幸いです。







 PAGE TOP
PAGE TOP